
哺乳動(dòng)物的DNA復(fù)制是在許多復(fù)制起點(diǎn)開始的,這些復(fù)制起點(diǎn)聚集在整個(gè)基因組的數(shù)千個(gè)復(fù)制域(RD)中垦巴。然而架独,目前尚不清楚每個(gè)RD內(nèi)的復(fù)制起源是隨機(jī)激活還是優(yōu)先在某些染色質(zhì)特征附近激活。
一.研究介紹
為了了解單個(gè)人類細(xì)胞中的DNA復(fù)制是如何在亞RD水平上調(diào)節(jié)的,作者使用STORM以超高分辨率直接可視化和定量表征跨S期的單個(gè)復(fù)制點(diǎn)(RFi)的時(shí)空組織蔫敲、形態(tài)和原位表觀遺傳特征添履,揭示了RFi傳播動(dòng)力學(xué)的分級(jí)徑向模式。
結(jié)合模擬和生物信息學(xué)分析票罐,指出了一種“CTCF組織的復(fù)制繁殖”(CoREP)模型叉趣,該模型表明早期S期亞RD水平復(fù)制激活的非隨機(jī)選擇機(jī)制,由CTCF有組織的染色質(zhì)結(jié)構(gòu)介導(dǎo)。這些發(fā)現(xiàn)為局部表觀遺傳環(huán)境在協(xié)調(diào)基因組DNA復(fù)制中的關(guān)鍵作用提供了重要的見解陶簿。
二.研究結(jié)果(節(jié)選)
在S期的特定階段欣骏,向細(xì)胞提供染料標(biāo)記的dUTP或EdU,將這些類似物分子結(jié)合到分裂期中DNA中航十,用STORM對(duì)標(biāo)記的RFi進(jìn)行成像耽晦,揭示了點(diǎn)狀分布以及跨S期,STORM(20 nm) 提供的卓越空間分辨率能夠?qū)Fi進(jìn)行更準(zhǔn)確的定量表征诊势,特別是對(duì)于基于數(shù)十納米內(nèi)多色共定位分析的后續(xù)發(fā)現(xiàn)洲棍。
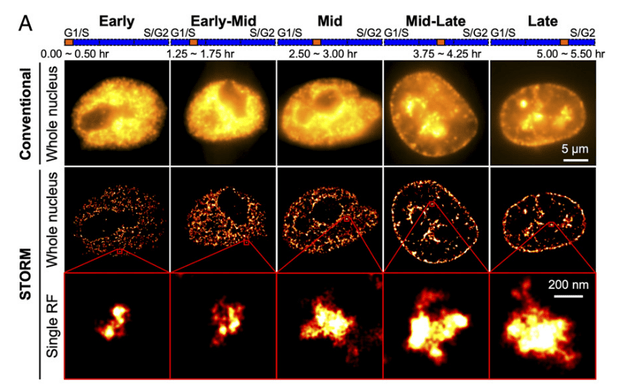 圖1. 跨S期RFi的STORM成像和定量表征
圖1. 跨S期RFi的STORM成像和定量表征
單個(gè)HeLa細(xì)胞核中RFi的常規(guī)和STORM圖像,在S期的五個(gè)不同階段(從左到右荔闭,每個(gè)階段有30分鐘的標(biāo)記)助碰。插圖(紅色框)顯示每個(gè)階段單個(gè)RFi 的放大代表性圖像。頂部的橙色條表示S期標(biāo)記周期的相對(duì)時(shí)間位置毒沥。
為了研究以亞衍射極限分辨率復(fù)制DNA的局部染色質(zhì)環(huán)境中的動(dòng)態(tài)變化婚乌,作者使用兩種光譜不同的染料Alexa 647和Cy3B原位共標(biāo)記RFi和與各種表觀遺傳特征相關(guān)的七個(gè)關(guān)鍵核標(biāo)記,并進(jìn)行雙色STORM成像以量化它們的共定位擒蝎。在這些標(biāo)記中中姜,已知CTCF通過形成染色質(zhì)環(huán)來調(diào)節(jié)3D染色質(zhì)結(jié)構(gòu),組蛋白修飾的H3K27ace和H3K4me3與轉(zhuǎn)錄活性染色質(zhì)相關(guān)跟伏,核層定位的層粘連蛋白 A/C主要與轉(zhuǎn)錄抑制的DNA 相關(guān)丢胚,而SUZ12構(gòu)成組蛋白H3的抑制復(fù)合物的一部分,增殖細(xì)胞核抗原(PCNA)是哺乳動(dòng)物復(fù)制的重要組成部分受扳,它與H2B一起作為陽性對(duì)照携龟。
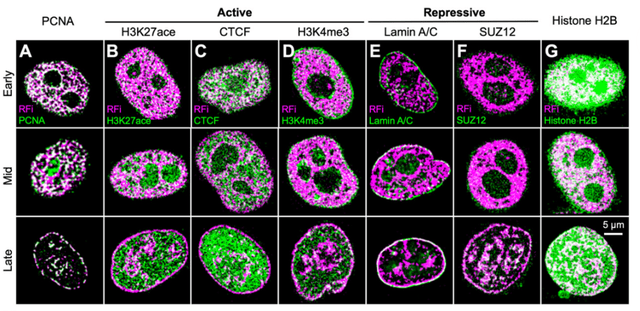 圖2. RFi相關(guān)表觀遺傳特征在S期原位的動(dòng)力學(xué)
圖2. RFi相關(guān)表觀遺傳特征在S期原位的動(dòng)力學(xué)
(A–G)RFi和七個(gè)關(guān)鍵核標(biāo)志物之間的共定位分析揭示RFi在整個(gè)S期的不同局部染色質(zhì)狀態(tài)。在S期早期(頂部)勘高,中期(中間)和晚期(底部)開始時(shí)標(biāo)記的新復(fù)制DNA(粉紅色)的細(xì)胞峡蟋,隨后用針對(duì)每個(gè)標(biāo)記物(綠色)的抗體進(jìn)行免疫標(biāo)記,并用雙色STORM成像华望。
為了探測(cè)單個(gè)RFi的空間組織和動(dòng)力學(xué)层亿,作者研究DNA復(fù)制在空間和時(shí)間上的傳播模式。為此立美,在S期三個(gè)不同階段的兩個(gè)連續(xù)30分鐘時(shí)間窗口內(nèi)用兩種光譜不同的染料Alexa 647和Atto 550標(biāo)記新復(fù)制的DNA匿又,此外,核層中的層粘連蛋白A/C用第三種染料Atto 488建蹄。在標(biāo)記細(xì)胞的多色STORM成像中稿屏,兩個(gè)時(shí)間窗口的相對(duì)空間分布揭示了RFi進(jìn)展的獨(dú)特時(shí)空模式。
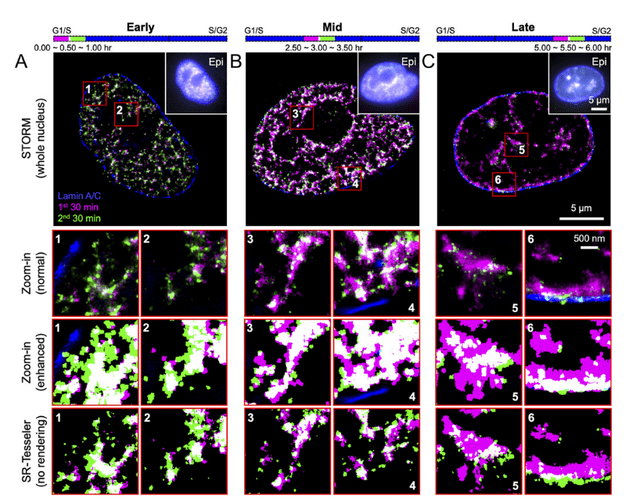 圖3. S期早期和晚期的RFi表現(xiàn)出相反的傳播動(dòng)力學(xué)時(shí)空模式
圖3. S期早期和晚期的RFi表現(xiàn)出相反的傳播動(dòng)力學(xué)時(shí)空模式
30分鐘窗口(紫色和綠色)內(nèi)新復(fù)制的DNA(紫色:第一個(gè)窗口;綠色:第二個(gè)窗口;白色:共定位RFi)與層粘連蛋白A/C(藍(lán)色)一起標(biāo)記每個(gè)細(xì)胞的核邊界饿严。頂部的紫色和綠色條表示S期標(biāo)記周期的相對(duì)時(shí)間位置滋冀。插圖(編號(hào)為1到6)顯示放大區(qū)域(紅色框)丙藤,顯示兩種顏色之間的正常對(duì)比度(頂部)和增強(qiáng)對(duì)比度(中),以獲得更好的可視化效果凿食。
為了理解RFi傳播獨(dú)特模式背后的機(jī)制基礎(chǔ)窘螃,CTCF通過形成染色質(zhì)環(huán)在調(diào)節(jié)3D基因組結(jié)構(gòu)中發(fā)揮的突出作用,作者使用RNA干擾物降低HeLa細(xì)胞中的CTCF表達(dá)馒俊,使用免疫染色在蛋白質(zhì)水平上證實(shí)了這一點(diǎn)册向,結(jié)果表明,CTCF及其介導(dǎo)的染色質(zhì)環(huán)結(jié)構(gòu)是RFi形態(tài)和傳播動(dòng)力學(xué)的關(guān)鍵調(diào)節(jié)因子避揍。
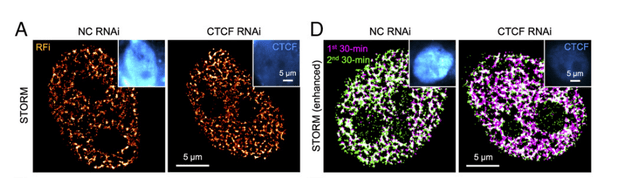 圖4. CTCF調(diào)節(jié)RFi形態(tài)和時(shí)空傳播動(dòng)力學(xué)
圖4. CTCF調(diào)節(jié)RFi形態(tài)和時(shí)空傳播動(dòng)力學(xué)
(A)用非特異性對(duì)照(NC)或CTCF siRNA轉(zhuǎn)染的細(xì)胞核中S期早期RFi的STORM圖像瘸卦。插圖(藍(lán)色)顯示了CTCF在RNA干擾下的成功下調(diào)。(D)在用NC或CTCF siRNA處理的細(xì)胞核的S期早期開始時(shí)蔓嚷,在兩個(gè)連續(xù)的30分鐘窗口(紫色和綠色)中標(biāo)記的新復(fù)制DNA的雙色STORM圖像柳锣。
總之,作者的研究為進(jìn)一步詢問DNA復(fù)制和其他相關(guān)核內(nèi)過程的時(shí)空組織和動(dòng)力學(xué)鋪平了道路雕蔽,為理解復(fù)制和轉(zhuǎn)錄之間的耦合提供了一個(gè)框架折柠,因?yàn)檗D(zhuǎn)錄活性啟動(dòng)子周圍的開放染色質(zhì)可以促進(jìn)附近復(fù)制起源的選擇,這可能反過來解釋轉(zhuǎn)錄活性基因傾向于早期復(fù)制的觀察結(jié)果批狐。
三.iSTORM預(yù)約試拍
目前液走,在國(guó)內(nèi),隨機(jī)光學(xué)重建顯微鏡STORM已成功實(shí)現(xiàn)商用贾陷,有需要STORM成像技術(shù)進(jìn)行實(shí)驗(yàn)研究的專家老師們,請(qǐng)文末填寫問卷嘱根,即可預(yù)約獲得 iSTORM 超高分辨率顯微成像系統(tǒng)試拍服務(wù)~
力顯智能現(xiàn)已發(fā)布的超高分辨率顯微成像系統(tǒng) iSTORM髓废,成功實(shí)現(xiàn)了光學(xué)顯微鏡對(duì)衍射極限的突破,使得在 20納米的分辨率尺度上從事生物大分子的單分子定位與計(jì)數(shù)该抒、亞細(xì)胞及超分子結(jié)構(gòu)解析慌洪、生物大分子生物動(dòng)力學(xué)等的研究成為現(xiàn)實(shí),從而給生命科學(xué)凑保、醫(yī)學(xué)等領(lǐng)域帶來重大突破冈爹。

超高分辨率顯微成像系統(tǒng) iSTORM 具有 20 納米超高分辨率、3通道同時(shí)成像欧引、3D同步拍攝枪蜕、實(shí)時(shí)重構(gòu)、2小時(shí)新手掌握等特點(diǎn)迈招,已實(shí)現(xiàn)活細(xì)胞單分子定位與計(jì)數(shù)农泊,并提供熒光染料選擇、樣本制備缘赋、成像服務(wù)與實(shí)驗(yàn)方案整體解決方案碰蚂,以納米級(jí)觀測(cè)精度惊派、高穩(wěn)定性、廣泛環(huán)境適用竣楼、快速成像送服、簡(jiǎn)易操作等優(yōu)異特性,獲得了超過50家科研小組和100多位科研人員的高度認(rèn)可亲堂。歡迎預(yù)約試拍炼杉!
參考文獻(xiàn)
References



